안녕하세요.
이번 글에서는 python에서 대표적인 차원 축소 알고리즘 중 하나인
umap을 통해서 차원 축소를 해보고 시각화로 결과를 살펴보는 예제를 다루어 보겠습니다.
모듈 설치
UMAP 시각화를 위해서는 umap-learn 모듈의 설치가 필요합니다.
다음의 명령어로 쉽게 설치가 가능합니다.
!pip install umap-learn
데이터셋 로드 및 정규화
이해를 돕기 위하여, 사이킷런에서 제공하는 iris 데이터셋을 대상으로 차원 축소 및 시각화를 해보겠습니다.
먼저, 필요한 모듈들을 import하고 데이터셋 로드 및 정규화를 해보겠습니다.
umap 차원 축소 시에는 정규화 과정이 꼭 필요하니 참고하세요.
# 0) 모듈 임포트
from sklearn.datasets import load_iris
from sklearn.preprocessing import StandardScaler
from sklearn.manifold import trustworthiness
from sklearn.metrics import silhouette_score
import pandas as pd
import numpy as np
import umap.umap_ as umap
import matplotlib.pyplot as plt
# 1) 데이터셋 로드
iris = load_iris()
df = pd.DataFrame(data=np.c_[iris.data], columns=['sepal length', 'sepal width', 'petal length', 'petal width'])
df['target'] = iris.target
# 2) 정규화 (feature만 표준화; target은 제외)
scaler = StandardScaler()
X_scaled = scaler.fit_transform(df.iloc[:, :-1])
y = df['target'].values
UMAP 차원 축소
umap 차원 축소 기법으로 기존에 'sepal length', 'sepal width', 'petal length', 'petal width'로
총 4가지였던 feature의 갯수를 2개로 줄이는 차원 축소를 해보겠습니다.
예시 코드 스니펫은 다음과 같습니다.
# 3) UMAP으로 2차원 임베딩
# - n_neighbors: 국소 구조를 얼마나 넓게 볼지(작을수록 미세한 구조), 기본 15
# - min_dist: 점들이 얼마나 조밀하게 모일지(작을수록 더 뭉침), 기본 0.1
# - metric: 거리 척도, 기본 'euclidean'
# - random_state: 재현성
umap_model = umap.UMAP(n_components=2, n_neighbors=15, min_dist=0.1, metric='euclidean', random_state=42)
embedding = umap_model.fit_transform(X_scaled)
df_umap = pd.DataFrame(embedding, columns=['UMAP-1', 'UMAP-2'])
df_umap['target'] = y
그러면 기존에 다음과 같이 4가지 feature를 가졌던 데이터프레임이

다음과 같이 2개의 feature로 차원 축소가 진행된 점을 확인 할 수 있습니다.

UMAP 설명력 지표 측정
umap 차원 축소 이후, 축소된 차원의 feature들이 기존 feature의 특징을
얼마나 잘 설명하고 있는지에 대한 지표를 측정할 수 있습니다.
이들은 보통 trustworthiness나 sihoutte score 등으로 평가가 가능한데요.
이들의 의미는 다음과 같습니다.
- Trustworthiness: 원공간 이웃관계가 임베딩에서 얼마나 잘 보존되었는지(1에 가까울수록 좋음)
- Silhouette score: 레이블(클래스)이 있다면, 임베딩 상에서 군집 분리가 얼마나 잘 되었는지를 간단히 확인
이 지표들을 측정하는 코드 예시는 다음과 같습니다.
# 4) 차원 축소 성능 지표 측정
tw = trustworthiness(X_scaled, embedding, n_neighbors=5)
sil = silhouette_score(embedding, y)
print(f"Trustworthiness (k=5): {tw:.4f}") # Trustworthiness (k=5): 0.9852
print(f"Silhouette score (on UMAP embedding): {sil:.4f}") # Silhouette score (on UMAP embedding): 0.5314
UMAP 차원 축소 결과 시각화
마지막으로, umap으로 차원 축소된 결과를 기반으로 시각화를 진행해 보겠습니다.
시각화 코드 및 결과 예시는 다음과 같습니다.
# 5) 시각화 (클래스별 산점도)
df0 = df_umap[df_umap['target'] == 0]
df1 = df_umap[df_umap['target'] == 1]
df2 = df_umap[df_umap['target'] == 2]
plt.figure(figsize=(6, 5))
plt.scatter(df0['UMAP-1'], df0['UMAP-2'], color='orange', alpha=0.7, label='setosa')
plt.scatter(df1['UMAP-1'], df1['UMAP-2'], color='red', alpha=0.7, label='versicolor')
plt.scatter(df2['UMAP-1'], df2['UMAP-2'], color='green', alpha=0.7, label='virginica')
plt.xlabel('UMAP-1')
plt.ylabel('UMAP-2')
plt.legend()
plt.title('Iris – UMAP(2D) embedding')
plt.show()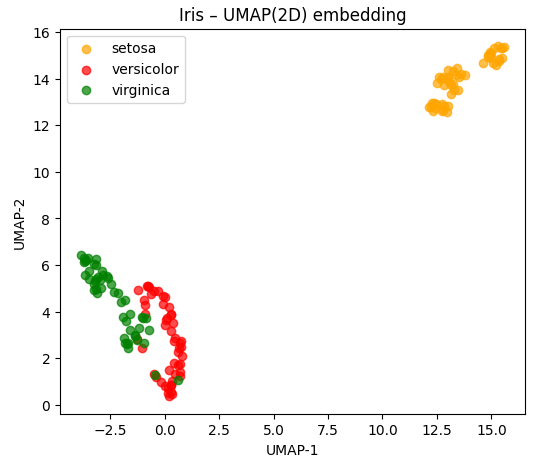
차원 축소된 두 개의 feature가 각각 x, y축으로 나타내졌으며,
세 집단이 비교적 어느 정도는 잘 구분되는 것을 산점도 상에서 확인이 가능했습니다.
이 글이 umap 차원 축소 과정에 도움이 되셨기를 바라겠습니다.
감사합니다.
'Python > Sklearn' 카테고리의 다른 글
| 테이블 도메인 딥러닝 예측 모델 TabPFN 파이썬 예제 (0) | 2025.09.09 |
|---|---|
| 파이썬 SMOTE 알고리즘 데이터 불균형 해결 예제 (0) | 2022.12.05 |
| [Sklearn] 파이썬 모델 앙상블 : 배깅 / 부스팅 / 보팅 함수 정리 (1) | 2022.09.25 |